Mise à jour des collections Ansible avec Renovate
Publié le :

Dans la démarche DevOps, on parle bien d’automatisation pour éliminer les tâches chronophages et sans valeurs ajoutées pour pouvoir se concentrer sur celles qui donnent de la valeur à notre produit.
C’est dans cette optique que j’ai mis en place l’automatisation des tests de mise à jour des rôles et des dépendances de type python (ansible, ansible-lint, molecule, …) dans mes collections Ansible. Pourquoi j’utilise des collections Ansible ? Tout simplement pour rationaliser le code (éviter la répétition de code), mais aussi pour gérer efficacement les versions de toutes mes dépendances. J’utilise de multiples collections en les limitant à de simples objectifs :
- Faire la configuration de base des serveurs qui viennent d’être provisionné que ce soit avec Packer ou appliqué directement sur les serveurs.
- Faire l’installation d’un composant : nginx, varnish, apache2, java, python…
- Monter un projet en utilisant au maximum les collections plutôt que des rôles
- …
Ces collections utilisent toutes des rôles réalisant des tâches assez basiques et qui sont correctement versionnés. Je déconseille fortement d’encapsuler des rôles dans d’autres rôles et puis dans d’autres… A moins d’adorer passer des heures à chercher la source de l’erreur qui vous tombe dessus.
Structure des Groupes dans Gitlab
Je suis hyper maniaque et je passe mon temps à ranger mes affaires. Et c’est vrai aussi sous Gitlab. J’ai créé une arborescence Ansible me permettant de retrouver rapidement un rôle, une collection, un projet contenant un inventaire et quelques playbooks …
ansible├── collections│ └── base├── images-docker│ └── molecule-collections├── projets│ ├── projet1-preprod│ └── projet1-prod└── roles ├── bootstrap └── motdJ’utilise la même arborescence sur mon pc, la seule différence se fait au niveau
des collections ou le dossier se nomme ansible_collections et contenant un
sous-dossier du nom de votre namespace. Cela afin que les commandes
ansible-galaxy fonctionnent. Je vous renvoie à mon explication sur les
collections Ansible
ansible├── ansible_collections│ └── stephrobert│ └── baseÉcriture de notre collection Ansible
Pour mon exemple, je vais créer une collection ansible permettant de configurer un serveur avec tout ce qu’il faut.
Création de la structure de la collection
Créons notre collection base qui doit se trouver dans le répertoire
ansible_collections/<namespace>:
mkdir -p ansible/ansible_collectionscd ansible/ansible_collectionsansible-galaxy collection init stephrobert.basecd stephrobert/baselstotal 20Kdrwxrwxr-x 2 vagrant vagrant 4,0K nov. 5 14:36 docs-rw-rw-r-- 1 vagrant vagrant 2,6K nov. 5 14:36 galaxy.ymldrwxrwxr-x 2 vagrant vagrant 4,0K nov. 5 14:36 plugins-rw-rw-r-- 1 vagrant vagrant 84 nov. 5 14:36 README.mddrwxrwxr-x 2 vagrant vagrant 4,0K nov. 5 14:36 rolesMaintenant initialisons la structure de molecule
molecule init scenario -d docker --provisioner-name ansible --verifier-name testinfraVous devriez obtenir cette structure :
molecule└── default ├── converge.yml ├── molecule.yml └── tests ├── conftest.py ├── __pycache__ │ ├── conftest.cpython-310.pyc │ └── test_default.cpython-310.pyc └── test_default.pyCréons quelques répertoires pour stocker nos playbooks, templates et nos fichiers :
mkdir {playbooks,files,templates}Configuration de la collection Ansible
La configuration se fait via le fichier galaxy.yml :
namespace: stephrobertname: baseversion: 1.0.1readme: README.mdauthors:- Stéphane ROBERT robert.stephane.28@gmail.comdescription: Install and configure Gitlab Runnerlicense:- GPL-2.0-or-laterlicense_file: ''
dependencies: community.general: ">=5.5.0"repository: https://gitlab.com/ansible-stephrobert/collections/gitlab-runner.gitdocumentation: /post/ansible-collection-molecule/homepage: /issues: https://gitlab.com/ansible-stephrobert/collections/gitlab-runner/-/issuesbuild_ignore: - galaxy.yml - .gitignoreLa première partie est déjà configurée, il faut juste éditer les champs du dépôt, les collections en dépendances, de la documentation, des issues et des fichiers qui devront être exclues de l’archive de la collection.
Vous pouvez déjà lancer la commande de build de la collection pour en vérifier le contenu.
tar tvfz stephrobert-base-1.0.0.tar.gz-rw-r--r-- 0/0 985 2022-11-05 15:21 MANIFEST.json-rw-r--r-- 0/0 2778 2022-11-05 15:21 FILES.json-rw-r--r-- 0/0 598 2022-11-05 14:42 .yamllintdrwxr-xr-x 0/0 0 2022-11-05 14:36 plugins/-rw-r--r-- 0/0 963 2022-11-05 14:36 plugins/README.mddrwxr-xr-x 0/0 0 2022-11-05 14:36 roles/drwxr-xr-x 0/0 0 2022-11-05 14:36 docs/drwxr-xr-x 0/0 0 2022-11-05 14:48 templates/drwxr-xr-x 0/0 0 2022-11-05 14:48 files/drwxr-xr-x 0/0 0 2022-11-05 14:42 molecule/drwxr-xr-x 0/0 0 2022-11-05 14:42 molecule/default/-rw-r--r-- 0/0 208 2022-11-05 14:42 molecule/default/molecule.ymldrwxr-xr-x 0/0 0 2022-11-05 14:42 molecule/default/tests/-rw-r--r-- 0/0 650 2022-11-05 14:42 molecule/default/tests/conftest.py-rw-r--r-- 0/0 217 2022-11-05 14:42 molecule/default/tests/test_default.py-rw-r--r-- 0/0 129 2022-11-05 14:42 molecule/default/converge.yml-rw-r--r-- 0/0 0 2022-11-05 15:16 requirements.ymldrwxr-xr-x 0/0 0 2022-11-05 14:48 playbooks/-rw-r--r-- 0/0 84 2022-11-05 14:36 README.mdConfiguration de molecule avec une image systemd
Nous allons utiliser un conteneur à base de debian11 permettant d’utiliser
systemd. Pourquoi ? Pour tout simplement permettre de démarrer des services
comme sur des VM.
Copier ce contenu dans votre fichier molecule/default/molecule.yml :
dependency: name: galaxy
driver: name: docker
platforms: - name: debian11_base image: debian:11 dockerfile: Dockerfile
privileged: True pre_build_image: False volumes: - "/sys/fs/cgroup:/sys/fs/cgroup:rw" - "/var/run/docker.sock:/var/run/docker.sock:rw" stop_signal: "SIGRTMIN+3" capabilities: - SYS_ADMIN - SYS_TIME - LINUX_IMMUTABLE - CAP_NET_BIND_SERVICE command: "/lib/systemd/systemd"
provisioner: name: ansible env: ANSIBLE_CONFIG: ../../ansible.cfg ANSIBLE_FORCE_COLOR: "True" options: v: True
verifier: name: testinfra options: v: True s: True
lint: | set -e yamllint . ansible-lint...Pour éviter de multiplier les fichiers indiquant les rôles en dépendances, je le
place à la racine de ma collection. Je crée ensuite dans le répertoire
molecule/default un lien pointant dessus :
---roles: - name: bootstrap src: https://github.com/stephrobert/ansible-role-bootstrap.git scm: git version: 1.0.0Vous remarquerez que j’utilise le scm de type git. Tout simplement pour pouvoir
renommer mon rôle en bootstrap plutôt qu’en stephrobert.bootstrap. Sinon le rôle
sera inaccessible depuis l’extérieur. Là pour y accéder, il faudra indiquer
steprobert.base.bootsrap dans votre playbook.
cd molecule/defaultln -s ../../requirements.ymlNotre image est construite avec ce Dockerfile qui doit se trouver aussi dans
le répertoire molecule/default :
FROM debian:11
RUN apt update -y && \ apt install -y --no-install-recommends systemd systemd-cron python3 python3-pip sudo bash vim ca-certificates && \ rm -Rf /usr/share/doc && \ rm -Rf /usr/share/man && \ apt clean && \ rm -rf /var/lib/apt/lists/* /tmp/* /var/tmp/*
RUN rm -f /lib/systemd/system/multi-user.target.wants/* \ /etc/systemd/system/*.wants/* \ /lib/systemd/system/local-fs.target.wants/* \ /lib/systemd/system/sockets.target.wants/*udev* \ /lib/systemd/system/sockets.target.wants/*initctl* \ /lib/systemd/system/sysinit.target.wants/systemd-tmpfiles-setup* \ /lib/systemd/system/systemd-update-utmp*
# Disable requiretty.RUN sed -i -e 's/^\(Defaults\s*requiretty\)/#--- \1/' /etc/sudoers
# Create `ansible` user with sudo permissionsENV ANSIBLE_USER=ansible SUDO_GROUP=sudoRUN set -xe && \ groupadd -r ${ANSIBLE_USER} && \ useradd -m -g ${ANSIBLE_USER} ${ANSIBLE_USER} && \ usermod -aG ${SUDO_GROUP} ${ANSIBLE_USER} && \ sed -i "/^%${SUDO_GROUP}/s/ALL\$/NOPASSWD:ALL/g" /etc/sudoers
# Custom bashrcRUN printf "# .bashrc \n\n\ alias pip=pip3 \n\ alias python=python3 \n\ alias ll='ls -l --color=auto' \n\ alias rm='rm -i' \n\ alias cp='cp -i' \n\ alias mv='mv -i' \n\n\ if [ -f /etc/bashrc ]; then \n\ . /etc/bashrc \n\ fi \n\ " > /root/.bashrc
VOLUME [ "/sys/fs/cgroup", "/tmp", "/run" ]ADD https://raw.githubusercontent.com/gdraheim/docker-systemctl-replacement/master/files/docker/systemctl3.py /usr/bin/systemctlRUN chmod +x /usr/bin/systemctlCMD [ "/lib/systemd/systemd" ]Vérifions que tout fonctionne :
molecule destroy # pour nettoyer les lancements précédentsmolecule convergeVous devriez au bout de quelques minutes obtenir un conteneur tournant en tache de fond :
docker psCONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES12b1ebbb23df molecule_local/debian:11 "/lib/systemd/systemd" 7 minutes ago Up 7 minutes debian11_basePour vous connecter dessus, il suffit de taper une des commandes suivantes :
molecule login# oudocker exec -it debian11_base bashroot@debian11base:/#Pour le moment le rôle ne s’est pas lancé puisque je n’ai pas encore créé mon playbook.
Ecriture du playbook
Nous allons écrire un playbook qui sera utilisé par molecule mais aussi lors de l’utilisation de la collection.
Dans le dossier playbooks créer un fichier bootstrap.yml et déposer ce contenu :
---- name: Bootstrap de notre machine hosts: all roles: - role: bootstrap vars: bootstrap_timeout: 10Pour que molecule fasse appel à notre playbook, il faut éditer le fichier converge.yml et remplacer le contenu par ceci
---- name: Import du playbook bootstrap ansible.builtin.import_playbook: ../../playbooks/bootstrap.ymlRelancez molecule et vous devriez voir l’installation des packages nécessaires au bon fonctionnement d’Ansible sur ma machine cible.
Ajoutons un autre rôle permettant de créer notre user admuser. Pour cela, je vais
utiliser un rôle en provenance de ma boutique claranet.users. Editez le
fichier requirements.yml et remplacez avec ceci :
---roles: - name: stephrobert.bootstrap version: 1.0.0 - name: claranet.users version: 2.0.0Modifions le playbook bootstrap.yml pour utiliser ce role :
---- name: Bootstrap de notre machine hosts: all roles: - role: bootstrap vars: bootstrap_timeout: 10 - role: claranet.users vars: users_lang: "en_US.UTF-8" users_packages: ["bash", "bash-completion", "vim", "sudo", "procps", "htop"] users: admuser: bashrc: - export PATH="$HOME/.local/bin:/sbin:/usr/sbin:$PATH" groups: - adm - sudo - root password: '{{ omit }}'Je vais en rester là, mais vous pourriez ajouter l’installation de sshd, de motd, de faire un peu d’hardening…
Ecriture des tests avec testinfra
On m’a demandé pourquoi j’utilise testinfra et pas Ansible pour faire mes tests ? Pour deux raisons :
- Je préfère utiliser un autre système que celui qui fait la configuration
- Je trouve plus simple d’écrire des tests en python qu’en Ansible.
Dans le dossier molecule/default/tests éditez le fichier test_default.py et
déposez-y ce contenu :
"""Role testing files using testinfra."""
def test_root_user(host): user = host.user("root") assert user.exists assert user.shell == "/bin/bash" assert user.home == f"/{user.name}" assert user.group == "root"
def test_root_profile_file(host): user_name = "root" file_name = f"/{user_name}/.profile" file = host.file(file_name) assert file.exists assert file.is_file assert file.user == "root" assert file.group == "root" assert file.mode == 0o644 assert file.contains("\nreadonly HISTFILE\n") assert file.contains("\nexport SHELL=/bin/bash\n") assert file.contains("\nexport LANG=fr_FR.UTF8\n")
def test_admuser_user(host): user = host.user("admuser") assert user.exists assert user.shell == "/bin/bash" assert user.home == f"/home/{user.name}" assert user.group == "admuser" assert "adm" in user.groups
def test_admuser_profile_file(host): user_name = "admuser" file_name = f"/home/{user_name}/.profile" file = host.file(file_name) assert file.exists assert file.is_file assert file.user == "root" assert file.group == "root" assert file.mode == 0o644 assert file.contains("\nreadonly HISTFILE\n") assert file.contains("\nexport SHELL=/bin/bash\n")
def test_installed_packages(host): assert host.package("bash").is_installed assert host.package("bash-completion").is_installed if str(host.system_info.distribution).lower() in ("centos", "redhat", "amzn"): assert host.package("vim-enhanced").is_installed else: assert host.package("vim").is_installed assert host.package("e2fsprogs").is_installed
def test_ssh_admuser(host): file_name = f"/home/admuser/.ssh" file = host.file(file_name) assert file.exists assert file.is_directory assert file.user == "admuser" assert file.group == "admuser" assert file.mode == 0o700On lance les tests avec la commande molecule verify :
molecule verify...
INFO Running default > verifyINFO Executing Testinfra tests found in /home/vagrant/Projets/personal/ansible/ansible_collections/stephrobert/base/molecule/default/tests/...============================= test session starts ==============================platform linux -- Python 3.10.6, pytest-7.1.3, pluggy-1.0.0 -- /usr/bin/python3rootdir: /home/vagrantplugins: testinfra-6.8.0collecting ... collected 6 items
molecule/default/tests/test_default.py::test_root_user[ansible:/debian11_base] PASSEDmolecule/default/tests/test_default.py::test_root_profile_file[ansible:/debian11_base] PASSEDmolecule/default/tests/test_default.py::test_admuser_user[ansible:/debian11_base] PASSEDmolecule/default/tests/test_default.py::test_admuser_profile_file[ansible:/debian11_base] PASSEDmolecule/default/tests/test_default.py::test_installed_packages[ansible:/debian11_base] PASSEDmolecule/default/tests/test_default.py::test_ssh_admuser[ansible:/debian11_base] PASSED
============================== 6 passed in 4.26s ===============================INFO Verifier completed successfully.Tous nos tests fonctionnent. Je rappelle que j’aurai dû faire du TDD en créant les tests puis en écrivant le code Ansible permettant de les résoudre.
Bon notre collection est prête à être envoyé sur notre dépôt, mais avant, attachons-nous à créer notre image Docker qui sera utilisé dans le Pipeline CI.
Construction de l’image Docker molecule
Dans cette image je vais mettre tout ce qui va permettre d’utiliser molecule : docker, ansible, ansible-lint, testinfra …
FROM ubuntu:22.04WORKDIR /COPY requirements.txt .ADD https://raw.githubusercontent.com/fsaintjacques/semver-tool/master/src/semver /usr/local/bin/semver
RUN chmod +x /usr/local/bin/semver && \ apt update && \ DEBIAN_FRONTEND=noninteractive apt install --no-install-recommends --assume-yes \ apt-transport-https \ ca-certificates \ lsb-release \ python3-pip \ curl \ ssh \ git \ software-properties-common && \ echo "deb [arch=amd64] https://download.docker.com/linux/ubuntu $(lsb_release -cs) stable" > /etc/apt/sources.list.d/docker.list && \ curl -fsSL https://download.docker.com/linux/ubuntu/gpg -o /etc/apt/trusted.gpg.d/docker.asc && \ apt update && \ DEBIAN_FRONTEND=noninteractive apt install --no-install-recommends --assume-yes docker-ce && \ pip install --no-cache-dir -r requirements.txt && \ apt autoremove --assume-yes && \ apt clean && \ rm --force --recursive /var/lib/apt/lists/* /tmp/* /var/tmp/*Le contenu des dépendances python requirements.txt :
ansible==6.5.0ansible-lint==6.8.2molecule-docker==1.1.0docker==6.0.0molecule==4.0.2pytest==7.2.0testinfra==6.0.0shyaml==0.6.2Ecriture d’une simple CI Gitlab
Pour notre pipeline d’intégration continue, nous allons utiliser trois étapes (stages gitlab) :
- Une étape de build
- Une étape de test : en fait le scenario test intègre tout
- Une étape de stockage de notre artefact (le tar.gz) sur Ansible Galaxy
En voici le contenu :
------stages: - build - tests - artefacts
workflow: rules: - if: '$CI_PIPELINE_SOURCE == "merge_request_event"' - if: '$CI_COMMIT_BRANCH == $CI_DEFAULT_BRANCH'services: - docker:dind
variables: COLLECTIONS_PATH: ${CI_PROJECT_DIR} COLLECTION_FACTS_PATH: ${COLLECTIONS_PATH}/ansible_collections/stephrobert/base
cache: &global_cache key: ${CI_COMMIT_REF_SLUG} paths: - ${COLLECTION_FACTS_PATH} policy: pull
build: stage: build cache: <<: *global_cache policy: push before_script: [] script: - chmod 755 . - ansible-galaxy role install -r ./requirements.yml -p ./roles/ - ansible-galaxy collection build -f . - ansible-galaxy collection install *.tar.gz -p "${COLLECTIONS_PATH}"
artifacts: paths: - '*.tar.gz' expire_in: 1 month
sanity_test: stage: tests script: - cd ${COLLECTION_FACTS_PATH} - ansible-test sanity
integration_tests: stage: tests needs: - build dependencies: - build script: - chmod 755 . - ansible-galaxy collection install *.tar.gz --force - ansible-galaxy role install -r ./requirements.yml - molecule destroy - molecule converge - molecule destroy
push: rules: - if: $CI_COMMIT_BRANCH == "main" stage: artefacts before_script: [] script: - echo `cat galaxy.yml |shyaml get-value version`...Tout est, si vous voulez valider localement votre CI, vous pouvez utiliser gcil. C’est ce que j’ai fait pour éviter les nombreux allers/retours.
Avant de pousser notre projet, il faut installer et configurer le runner. De mon
côté, je possède déjà une instance de gitlab-runner sur une de mes machines de
mon homelab.
Ah oui, le stage push ne sera lancé que sur la branche main. Renovate va
créer des branches sur lequel les tests seront lancés, mais ce n’est que lorsque
le merge sera fait que le push s’effectuera.
Installation et Configuration du runner Gitlab
Dans un premier temps voyons comment installer un runner Gitlab-CI. Je vais utiliser un runner tournant sous Docker monté sur une machine Linux. Bien sûr, nous allons écrire et utiliser une collection ansible pour réaliser cette tâche.
Installation de Gitlab Runner
Je vais l’installer sur une Ubuntu 22.04, mais le script fonctionne pour la plupart des distributions :
sudo sucurl -L "https://packages.gitlab.com/install/repositories/runner/gitlab-runner/script.deb.sh" | bashapt install gitlab-runnergitlab-runner status
Runtime platform arch=amd64 os=linux pid=1017961 revision=0d4137b8 version=15.5.0gitlab-runner: Service is runningAjoutons le compte gitlab-runner aux sudoers sans mot de passe :
sudo visudoAjoutez cette ligne à la fin du fichier :
gitlab-runner ALL=(ALL) NOPASSWD: ALLEnregistrement du runner
Il faut se connecter avec le compte gitlab-runner .
sudo su gitlab-runnerModifions la configuration pour ajouter des volumes et le passer le priviliged à
true, ça se trouve dans la section runners.docker :
concurrent = 1check_interval = 0
[session_server] session_timeout = 1800
[[runners]] name = "docker-runner" url = "https://gitlab.com/" id = 18755596 token = "K4vP7yF8tkCid7p9BAJD" token_obtained_at = 2022-11-05T18:36:34Z token_expires_at = 0001-01-01T00:00:00Z executor = "docker" [runners.custom_build_dir] [runners.cache] [runners.cache.s3] [runners.cache.gcs] [runners.cache.azure] [runners.docker] tls_verify = false image = "docker:20.10.16" privileged = true disable_entrypoint_overwrite = false oom_kill_disable = false disable_cache = false volumes = ["/var/run/docker.sock:/var/run/docker.sock", "/cache"] shm_size = 0Enregistrer et relancer gitlab-runner :
sudo gitlab-runner restartEt finissons par ajouter le user gitlab-runner au groupe docker :
usermod -a -G docker gitlab-runnerReconnectez avec ce compte et tapez la commande docker ps. Si tout est ok,
vous verrez la liste des containers vide.
Premier build sur notre runner
Tout est prêt pour lancer notre premier push dans notre dépôt nouvellement créé. Adaptez les commandes avec l’url de votre dépôt :
git initgit config --global init.defaultBranch maingit branch -m maingit remote add origin git@gitlab.com:ansible-stephrobert/collections/base.gitgit add .git commit -m "Initial commit"\ngit push -u origin mainNos trois stages passent avec succès !!!
Testons Renovate
J’ai déjà documenté l’utilisation dans ce billet.
Je vais simplement ajouter les projets de l’image et de la collection :
module.exports = { hostRules: [ { hostType: "docker", matchHost: "registry.gitlab.com/dockerfiles6", username: process.env.DEPLOY_USER, password: process.env.DEPLOY_TOKEN, }, ], endpoint: "https://gitlab.com/api/v4/", platform: "gitlab", gitUrl: "https", onboardingConfig: { extends: ["config:base"], }, vulnerabilityAlerts: { labels: ["security"], schedule: "at any time", }, repositories: [ "dockerfiles6/template/ci-build", "dockerfiles6/images/hadolint", "dockerfiles6/images/ansible-lint", "dockerfiles6/images/lastversion", "dockerfiles6/images/container-structure-test", "dockerfiles6/images/img", "b4288/infra-as-code-homelab", "dockerfiles6/build-python-wheel", "ansible-stephrobert/collections/base", "dockerfiles6/images/molecule-collections" ],};Allez, on lance deux pipelines renovate ! Un pour configurer le dépôt pour
renovate et un second pour lancer réellement l’analyse des dépendances et
créez les branches en cas de maj disponibles.
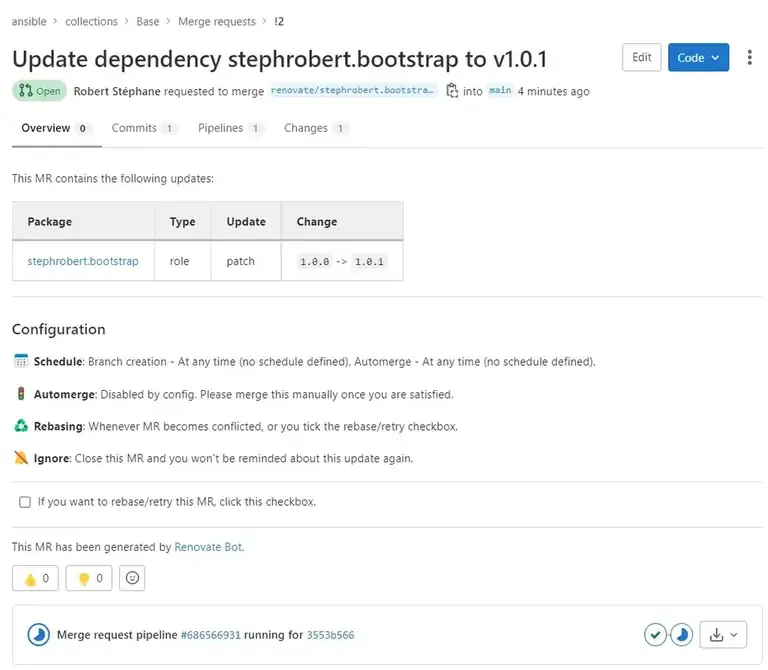
Cool, renovate a bien détecté l’existence d’une nouvelle version du rôle base.
Fini de devoir redescendre le projet pour simplement checker si la collection fonctionne toujours après la mise à jour des dépendances. Sur des dizaines de rôles et une dizaine de collections ça en fait des heures de gagner. En cas d’erreur, il faudra tout de même apporter les corrections. Par contre, avec toutes ses heures gagnées, vous pourrez les consacrer à faire votre veille techno, travailler sur des projets à plus fortes valeurs ajoutés.
Pour éviter d’avoir trop de branches créées par renovate, donc éviter de faire
trop de bruit, vous pouvez grouper toutes les mises à jour dans une seule merge
request. Pour cela, il faut ajouter une règle dans le fichier renovate.json
comme ceci :
{ "$schema": "https://docs.renovatebot.com/renovate-schema.json", "extends": ["config:base"], "packageRules": [ { "groupName": "all dependencies", "groupSlug": "all", "matchPackagePatterns": ["*"] } ]}Vous obtiendrez une seule merge request portant le nom chore(deps): update all dependencies.
Plus loin
Il ne reste plus qu’à écrire d’autres collections pour les midlleware en utilisant cette collection de base, des collections par projet utilisant la collection de base et celles des middlewares, à mettre en place renovate dessus. Après cela, vous pourrez gérer plus sereinement ceux-ci !
Vous pouvez retrouver tous les codes sources de ce projet dans les dépôts suivants :
